Browse Source
Revert "Retrait des fichiers de script et de données"
This reverts commit 036477883f.
master
9 changed files with 1815 additions and 1125 deletions
Split View
Diff Options
-
+38 -6README.md
-
BINREADME.png
-
+1583 -0README.svg
-
+4 -20cloture.Rmd
-
+136 -0donnees.R
-
+4 -0donnees.sh
-
+48 -21functionsOvalide.R
-
+0 -1078index.rmd
-
+2 -0render.sh
+ 38
- 6
README.md
View File
| @@ -5,32 +5,64 @@ | |||
| Les scripts ont été développés sous R version 3.3.1, à l'aide des packages suivants : | |||
| * Versions stables (CRAN) | |||
| * tidyverse | |||
| * tidyr 0.5.1 | |||
| * ggplot2 2.1.0 | |||
| * lubridate 1.5.6 | |||
| * rvest 0.3.2 | |||
| * stringr 1.0.0 | |||
| * readr 0.2.2 | |||
| * magrittr 1.5 | |||
| * dplyr 0.5.0 | |||
| * knitr 1.13 | |||
| * rmarkdown 1.0 | |||
| * htmlwidgets 0.7 | |||
| * Versions de développement (github) | |||
| * DT 0.1.57 | |||
| Ces paquets sont installés au niveau système pour tous les utilisateurs livenne. | |||
| ## Utilisation | |||
| Le rapport est sous la forme d'une application shiny+rmarkdown. | |||
| Une page permet d'entrer les informations nécessaires à la génération du rapport (codes d'accès, fichier d'exhaustivité issu de la requête BO), qui est généré au fur et à mesure dans un second onglet. | |||
| Une fois le rapport généré, il est possible d'en télécharger une version statique. | |||
| La génération du rapport est séparée en deux parties : | |||
| * l'acquisition et le pré-traitement des données | |||
| * la génération du rapport en lui-même | |||
| ### Acquisition des données | |||
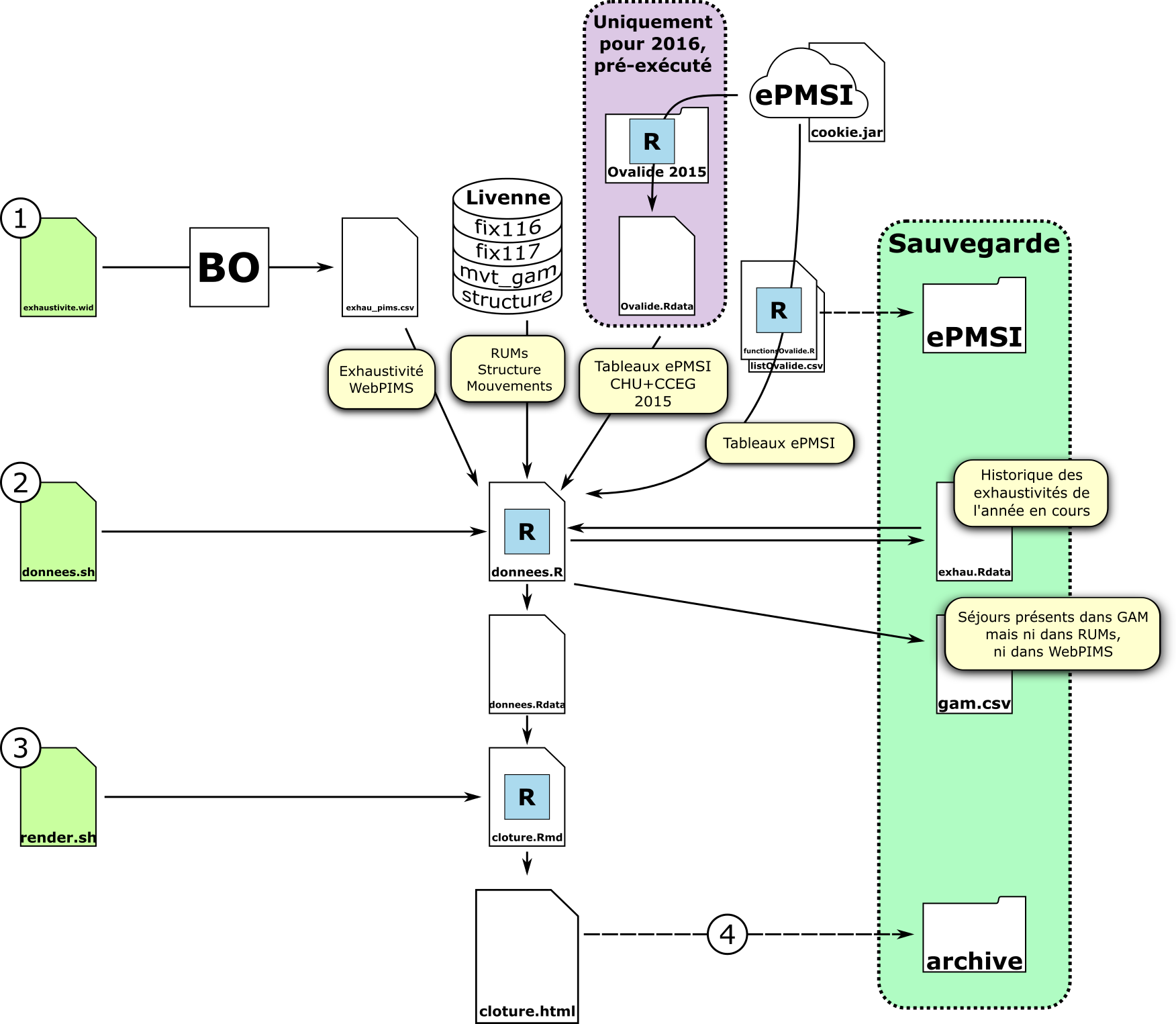
| Une première étape manuelle consiste à utiliser la requête Business Objects **exhaustivite.wid** présente dans le répertoire et de sauvegarder le fichier csv (choisir les options de format : encodage en UTF8, séparateur = ","). | |||
| Une première étape manuelle consiste à utiliser la requête Business Objects **exhaustivite.wid** présente dans le répertoire et de sauvegarder le fichier csv (choisir les options de format : encodage en UTF8, séparateur = ",") sous le nom **exhau_pims.csv** dans le répertoire du script. | |||
| Le script **donnees.sh** appelle le script **donnees.R** qui récupère les autres données depuis les différentes sources (base de données MySQL (tables fix116, fix117, mvt_gam et structure) sur livenne et données ePMSI sur le site de l'ATIH, exhaustivité WebBIMS dans le fichier **exhau_pims.csv**, tableaux ePMSI CCEG+CHU de 2015 pré-assemblés dans le fichier **Ovalide.Rdata** (nécessaires uniquement pour les bilans de 2016). | |||
| Les tableaux ePMSI sont archivés dans le dossier **ePMSI**. | |||
| Il nécessite un appel en ligne de commande avec les paramètres suivants : | |||
| * nom d'utilisateur de session CHU | |||
| * mot de passe de session CHU (pour passer à travers le proxy) | |||
| * nom d'utilisateur ePMSI | |||
| * mot de passe ePMSI (pour télécharger les tableaux depuis ePMSI) | |||
| * nom d'utilisateur MySQL livenne | |||
| * mot de passe MySQL livenne (pour interroger la base de données) | |||
| * annee de la clôture | |||
| * mois de la clôture | |||
| Un appel incorrect au script rappelle les paramètres de ligne de commande à utiliser. | |||
| Les noms d'utilisateurs/mots de passe contenant des caractères spéciaux (autres qu'alphanumériques) doivent être entourés de guillemets. | |||
| Le script **donnees.R** utilise le fichier **Ovalide.Rdata** contenant les données fusionnées du CHU et du CCEG pour 2015. Après 2016 ce fichier ne sera plus utile. Il utilise également le fichier **exhau.Rdata** pour sauvegarder les données d'exhaustivité depuis le début de l'année en cours. Ce fichier est réinitialisé en début d'année. | |||
| Les données récupérées et pré-traitées son enregistrées dans le fichier **donnees.Rdata** pour l'utilisation par le script de génération du rapport. | |||
| Le fichier **gam.csv** est généré. Il contient les séjours présents dans la base de mouvements (GAM), mais qui n'ont ni été envoyés à l'ATIH, ni sont connus comme étant non exhaustifs dans WebPIMS. Il s'agit principalement d'erreurs de la base GAM (annulations, transformations) à corriger. | |||
| ### Génération du rapport | |||
| Le fichier **cloture.html** généré peut être vérifié avant publication. La publication se fait en copiant le fichier dans le répertoire /var/www/html/cloture. | |||
| Le script **render.sh** appelle la commande *render* de Rmarkdown pour générer le rapport de clôture **cloture.html** à partir du fichier **cloture.Rmd**. | |||
| Celui-ci génère les différents tableaux et figures à la volée à partir des données pré-traitées fournies par **donnees.R** dans le fichier **donnees.Rdata**. | |||
| Le fichier **cloture.html** généré peut être vérifié avant publication. La publication se fait en copiant le fichier dans le répertoire au-dessus dans l'arborescence (/var/www/html/cloture). | |||
| Ce fichier doit également être copié dans le répertoire **archive** sous la forme **cloture*ANNEE_MOIS*.html** pour archivage. | |||
|  | |||
BIN
README.png
View File
+ 1583
- 0
README.svg
File diff suppressed because it is too large
View File
+ 4
- 20
cloture.Rmd
View File
| @@ -4,22 +4,15 @@ output: | |||
| html_document: | |||
| toc: true | |||
| toc_float: true | |||
| params: | |||
| annee: NA | |||
| mois: NA | |||
| rum: NA | |||
| rss: NA | |||
| exhau: NA | |||
| exhau_pims: NA | |||
| Ovalide: NA | |||
| OvalideP: NA | |||
| --- | |||
| ```{r init, echo = F, message = F} | |||
| library(tidyverse) | |||
| library(DT) | |||
| library(ggplot2) | |||
| library(knitr) | |||
| library(tidyr) | |||
| library(stringr) | |||
| library(dplyr) | |||
| library(plotly) | |||
| library(lubridate) | |||
| @@ -34,19 +27,10 @@ options(DT.options = list(paging = F, | |||
| info = F, | |||
| dom = "Bfrtip", | |||
| buttons = c("copy", "excel"))) | |||
| pdf(NULL) | |||
| ``` | |||
| ```{r data} | |||
| Ovalide <- params$Ovalide$tab | |||
| OvalideP <- params$OvalideP$tab | |||
| annee <- params$annee | |||
| mois <- params$mois | |||
| rum <- params$rum | |||
| rss <- params$rss | |||
| exhau <- params$exhau | |||
| exhau_pims <- params$exhau_pims | |||
| load("donnees.Rdata") | |||
| mois_label <- c("Janvier", | |||
| "Février", | |||
+ 136
- 0
donnees.R
View File
| @@ -0,0 +1,136 @@ | |||
| source("functionsOvalide.R") | |||
| library(lubridate) | |||
| library(RMySQL) | |||
| # Récupération des arguments pour utilisation non interactive ---- | |||
| arguments <- commandArgs() | |||
| if (length(arguments) != 13) | |||
| { | |||
| print ("Usage : donnees.sh CHUuser CHUpass ATIHuser ATIHpass LivenneUser LivennePass annee mois") | |||
| quit() | |||
| } else | |||
| { | |||
| CHUuser <- arguments[6] | |||
| CHUpass <- arguments[7] | |||
| ATIHuser <- arguments[8] | |||
| ATIHpass <- arguments[9] | |||
| user <- arguments[10] | |||
| password <- arguments[11] | |||
| annee <- as.numeric(arguments[12]) | |||
| mois <- as.numeric(arguments[13]) | |||
| } | |||
| # Récupération des fichiers ePMSI ---- | |||
| print("1/9 - Connexion à ePMSI") | |||
| connectOvalide(CHUuser, CHUpass, ATIHuser, ATIHpass) | |||
| print(str_c("2/9 - Téléchargement du tableau de", mois, annee, sep = " ")) | |||
| getOvalide(CHUuser, CHUpass, annee, mois) | |||
| print(str_c("3/9 - Téléchargement du tableau de", mois, annee - 1, sep = " ")) | |||
| getOvalide(CHUuser, CHUpass, annee - 1, mois) | |||
| # Extraction des données des fichiers ePMSI ---- | |||
| print("4/9 - Extraction des tableaux ePMSI") | |||
| read.csv2("listOvalide.csv") %>% | |||
| { | |||
| apply(., 1, . %>% | |||
| { | |||
| df <- extractOvalide(annee, mois, .[2], .[3]) | |||
| names(df) <- LETTERS[1:length(df)] | |||
| df | |||
| }) ->> Ovalide | |||
| names(Ovalide) <<- .$name | |||
| apply(., 1, . %>% | |||
| { | |||
| df <- extractOvalide(annee - 1, mois, .[2], .[3]) | |||
| names(df) <- LETTERS[1:length(df)] | |||
| df | |||
| }) ->> OvalideP | |||
| names(OvalideP) <<- .$name | |||
| } | |||
| # Récupération des données ePMSI de 2015 fusionnées CCEG-CHU ---- | |||
| if (annee == 2016) | |||
| { | |||
| load("Ovalide.Rdata") | |||
| OvalideP <- CCEGCHU2015[[str_c("mois", mois, sep = "")]] | |||
| rm("CCEGCHU2015") | |||
| } | |||
| # Nettoyage des fichiers extraits. Les archives sont conservées. ---- | |||
| unlink(str_c("OVALIDE T2A.MCO.DGF", annee, mois, "html", sep = ".")) | |||
| unlink(str_c("OVALIDE T2A.MCO.DGF", annee - 1, mois, "html", sep = ".")) | |||
| # RUM + structure + calcul durée de séjour ---- | |||
| print("5/9 - LIVENNE : RUM et structure") | |||
| src_mysql("pmsi_dim", "localhost", user = user, password = password) %>% | |||
| tbl(sql("SELECT finess, idhosp, idrss, idrum, date_entree, date_sortie, cmd, um FROM fix116 | |||
| UNION | |||
| SELECT finess, idhosp, idrss, idrum, date_entree, date_sortie, cmd, um FROM fix117")) %>% | |||
| collect(n = Inf) %>% | |||
| mutate(date_entree = date_entree %>% as.Date, | |||
| date_sortie = date_sortie %>% as.Date) %>% | |||
| left_join(src_mysql("pmsi_dim_nom", "localhost", user = user, password = password) %>% | |||
| tbl("structure") %>% | |||
| filter(um != "") %>% | |||
| select(uf_date_ferm, um, service_libelle, pole_libelle) %>% | |||
| arrange(um, uf_date_ferm) %>% | |||
| distinct %>% | |||
| collect(n = Inf) %>% | |||
| add_rownames(var = "dummy") %>% # Dédoublonnage : 1 um -> 1 couple libellés | |||
| group_by(um) %>% | |||
| filter(dummy == last(dummy)) %>% | |||
| select(-dummy, -uf_date_ferm) %>% | |||
| ungroup) %>% | |||
| mutate(idrss = ifelse(finess == "540000031", str_c("m", idrss), idrss)) %>% # maternité | |||
| mutate(idrss = ifelse(finess == "540020112", str_c("c", idrss), idrss)) %>% # CCEG | |||
| mutate(idhosp = idhosp %>% str_replace_all(" ", "")) %>% | |||
| mutate(duree_rum = date_sortie - date_entree) %>% # durée rum | |||
| left_join(group_by(., idrss) %>% | |||
| summarise(duree_rss = sum(duree_rum))) -> rum | |||
| # CLOS14 Hémato ---- | |||
| rum %<>% | |||
| mutate(pole_libelle = ifelse(pole_libelle == "CLOS14 HEMATOLOGIE", "SPECIALITES MEDICALES", pole_libelle), | |||
| service_libelle = ifelse(service_libelle == "CLOS14 HEMATO.S.INTENSIFS", "SERVICE HEMATOLOGIE", service_libelle)) | |||
| # Création des RSSs des 3 dernières années à partir des RUMs ---- | |||
| print("6/9 - Création des RSS") | |||
| rum %>% | |||
| group_by(idrss) %>% | |||
| filter(idrum == max(idrum)) %>% | |||
| ungroup %>% | |||
| mutate(annee_sortie = year(date_sortie), | |||
| mois_sortie = month(date_sortie)) %>% | |||
| filter(annee_sortie > annee - 3) -> rss | |||
| # Sélection uniquement des RUMs appartenant aux RSS ---- | |||
| print("7/9 - Sélection des RUMs") | |||
| rum %<>% | |||
| right_join(rss %>% select(idrss, annee_sortie, mois_sortie)) | |||
| # Exhaustivité ---- | |||
| print("8/9 - Exhaustivité") | |||
| exhau_pims <- read_csv("exhau_pims.csv") | |||
| names(exhau_pims) <- c("Pole", "Service", "RUM", "DP", "Erreur", "Sortie", "Duree", "RSS") | |||
| exhau_pims %<>% | |||
| mutate(Sortie = Sortie %>% as.Date) %>% | |||
| filter(month(Sortie) <= mois) | |||
| dbConnect(MySQL(), host = "localhost", dbname = "pmsi_dim", user = user, password = password) %>% | |||
| dbWriteTable("exhaustivite", data.frame(annee = annee, mois = mois, nb_rss_tot = exhau_pims %>% distinct(RSS, .keep_all = T) %>% nrow, nb_rss_manq = exhau_pims %>% filter(is.na(DP)) %>% distinct(RSS) %>% nrow), row.names = F, append = T) | |||
| an <- annee | |||
| src_mysql("pmsi_dim", "localhost", user = user, password = password) %>% | |||
| tbl("exhaustivite") %>% | |||
| collect(n = Inf) %>% | |||
| filter(annee == an) %>% | |||
| full_join(data.frame(mois = 1:12), by = "mois") %>% | |||
| select(annee, mois, rss = nb_rss_tot, manq = nb_rss_manq) -> exhau | |||
| # Écriture des données ---- | |||
| print("9/9 - Écriture des données") | |||
| save(Ovalide, OvalideP, annee, mois, rum, rss, exhau, exhau_pims, file = "donnees.Rdata") | |||
+ 4
- 0
donnees.sh
View File
| @@ -0,0 +1,4 @@ | |||
| #!/bin/bash | |||
| R --vanilla --quiet --slave --args $1 $2 $3 $4 $5 $6 $7 $8 < donnees.R | |||
+ 48
- 21
functionsOvalide.R
View File
| @@ -1,7 +1,7 @@ | |||
| library(tidyverse) | |||
| library(dplyr) | |||
| library(magrittr) | |||
| library(httr) | |||
| #library(RCurl) | |||
| library(RCurl) | |||
| library(readr) | |||
| library(stringr) | |||
| library(rvest) | |||
| @@ -45,53 +45,80 @@ extractOvalide <- function(annee, mois, table, subtable = "") | |||
| connectOvalide <- function(CHUuser, CHUpass, ATIHuser, ATIHpass) | |||
| { | |||
| curlopts <- list(proxy = str_c(CHUuser, ':', CHUpass, '@ssl-proxy.chu-nancy.fr:8080'), | |||
| follow = T, | |||
| cookiejar = 'cookie.jar', | |||
| cookiefile = 'cookie.jar') | |||
| curl <- str_c('curl -x "', CHUuser, ':', CHUpass, '@ssl-proxy.chu-nancy.fr:8080" -b cookie.jar -c cookie.jar -L ') | |||
| pasrel <- 'https://pasrel.atih.sante.fr/cas/login' | |||
| epmsi <- 'https://epmsi.atih.sante.fr/' | |||
| # Config proxy | |||
| set_config(use_proxy(url = "ssl-proxy.chu-nancy.fr", port = 8080, username = CHUuser, password = CHUpass)) | |||
| unlink("cookie.jar") | |||
| # Cookie | |||
| print("ePMSI : COOKIE") | |||
| system(str_c(curl, pasrel)) | |||
| # Token | |||
| print("ePMSI : TOKEN") | |||
| GET(pasrel) %>% | |||
| content %>% | |||
| getURL(pasrel, .opts = curlopts) %>% | |||
| read_html %>% | |||
| html_node("input[name='lt']") %>% | |||
| html_attr("value") -> token | |||
| print(token) | |||
| # Login | |||
| print("ePMSI : LOGIN") | |||
| POST(pasrel, body = list(username = ATIHuser, password = ATIHpass, lt = token, "_eventId" = "submit", submit = "SE+CONNECTER"), encode = "form") %>% cookies -> cookie | |||
| system(str_c(curl, '-d "username=', ATIHuser, '&password=', curlPercentEncode(ATIHpass), '<=', token, '&_eventId=submit&submit=SE+CONNECTER" ', pasrel)) | |||
| # Auth | |||
| print("ePMSI : AUTH") | |||
| GET(str_c(epmsi, 'authenticate.do'), set_cookies(cookie$value %>% setNames(cookie$name))) %>% cookies | |||
| system(str_c(curl, epmsi, 'authenticate.do')) | |||
| } | |||
| getOvalide <- function(CHUuser, CHUpass, annee, mois) | |||
| { | |||
| curlopts <- list(proxy = str_c(CHUuser, ':', CHUpass, '@ssl-proxy.chu-nancy.fr:8080'), | |||
| follow = T, | |||
| cookiejar = 'cookie.jar', | |||
| cookiefile = 'cookie.jar') | |||
| epmsi <- 'https://epmsi.atih.sante.fr/' | |||
| # Applis | |||
| print("ePMSI : APPLIS") | |||
| GET(str_c(epmsi, 'jsp/epmsi/applis/applis.jsp')) | |||
| getURL(str_c(epmsi, 'jsp/epmsi/applis/applis.jsp'), | |||
| .opts = curlopts) %>% | |||
| cat | |||
| # Ovalide | |||
| print("ePMSI : OVALIDE") | |||
| GET(str_c(epmsi, 'jsp/epmsi/applis/applisMat2a.jsp')) | |||
| getURL(str_c(epmsi, 'jsp/epmsi/applis/applisMat2a.jsp'), | |||
| referer = str_c(epmsi, 'jsp/epmsi/applis/applis.jsp'), | |||
| .opts = curlopts) %>% | |||
| cat | |||
| # Ovalide MCO T2A | |||
| print("ePMSI : MCO T2A") | |||
| GET(str_c(epmsi, 'appli_16.do?champPmsi=1&statut=1&applicationType=3')) | |||
| getURL(str_c(epmsi, 'appli_16.do?champPmsi=1&statut=1&applicationType=3'), | |||
| referer = str_c(epmsi, 'jsp/epmsi/applis/applisMat2a.jsp'), | |||
| .opts = curlopts) %>% | |||
| cat | |||
| # Resultats pour annee/mois | |||
| print("ePMSI : RESULTATS") | |||
| GET(str_c(epmsi, 'appli_05.do?year=', annee, '&period=', mois)) | |||
| getURL(str_c(epmsi, 'appli_05.do?year=', annee, '&period=', mois), | |||
| referer = str_c(epmsi, 'appli_16.do?champPmsi=1&statut=1&applicationType=3'), | |||
| .opts = curlopts) %>% | |||
| cat | |||
| # Tableaux | |||
| print("ePMSI : TABLEAUX") | |||
| GET(str_c(epmsi, 'appli_05.zip?action=4&win=1')) %>% | |||
| content %>% | |||
| writeBin(con = str_c('ePMSI/', annee, '_', mois, '.zip')) | |||
| getBinaryURL(str_c(epmsi, 'appli_05.zip?action=4&win=1'), | |||
| referer = str_c(epmsi, 'appli_05.do?year=', annee, '&period=', mois), | |||
| .opts = curlopts) %>% | |||
| writeBin(con = str_c('ePMSI/', annee, '_', mois, '.zip')) | |||
| unzip(str_c('ePMSI/', annee, '_', mois, '.zip')) | |||
| } | |||
+ 0
- 1078
index.rmd
File diff suppressed because it is too large
View File
+ 2
- 0
render.sh
View File
| @@ -0,0 +1,2 @@ | |||
| #!/bin/bash | |||
| R --vanilla -e 'rmarkdown::render("cloture.Rmd")' | |||